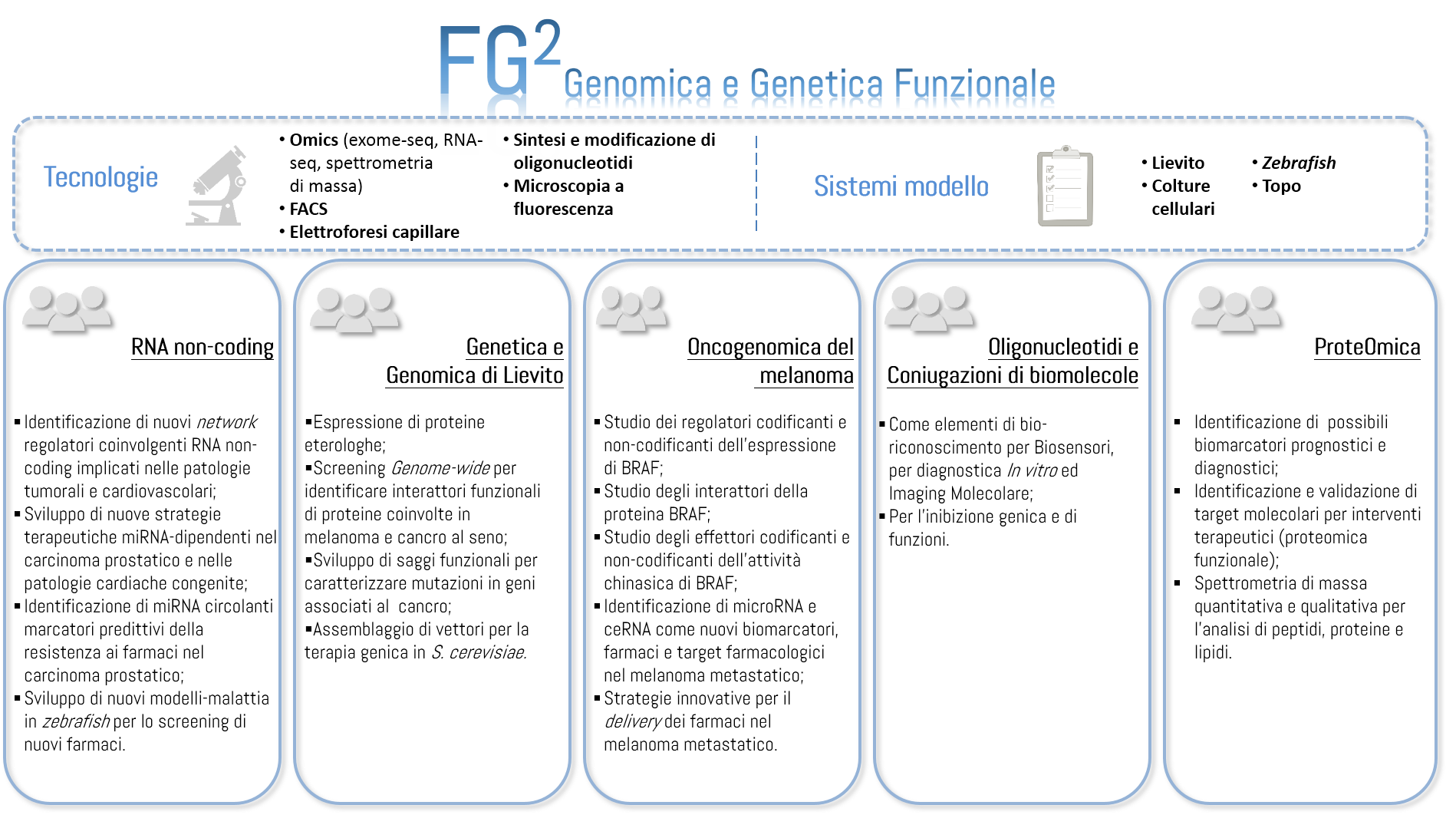
Gli obiettivi finali del Laboratorio di genetica e genomica Funzionale (FG2) sono l'identificazione e la caratterizzazione di mutazioni e altri fattori regolatori che possono essere coinvolti nello sviluppo e nella progressione della malattia. Le malattie più comuni, come quelle cardiovascolari e il cancro, sono causate da una combinazione di multipli fattori genetici e ambientali. La comprensione della base molecolare della patologia, e dell’interazione dei fattori genetici, indirizza verso una migliore prevenzione, diagnosi e trattamento. Inoltre, accrescere la conoscenza di questi fattori e delle loro interrelazioni, mediante studi funzionali, può portare allo sviluppo di nuove strategie di trattamento e prevenzione.
RNA non-coding
Un elevato numero di malattie cardiache è causato da alterazioni genetiche che influenzano lo sviluppo e/o l’attività cardiaca.
Il gruppo di ricerca utilizza zebrafish come sistema modello per l’identificazione di geni/meccanismi che regolano il normale sviluppo e la funzionalità cardiache con particolare attenzione al coinvolgimento dei microRNA (miRNA) come elementi cruciali di regolazione. L’utilizzo di zebrafish rispetto ai classici sistemi modello presenta numerosi vantaggi: alto grado di omologia con l’uomo, elevata fertilità, fecondazione esterna, rapido sviluppo, estrema semplicità di manipolazione genetica. Dal gruppo di lavoro IFC sono stati già identificati i miRNA che sono alterati in patologie cardiache congenite, caratterizzandone i loro target. Gli studi funzionali di guadagno e perdita di funzione in zebrafish consentono di valutare il loro potenziale impiego come nuovi target terapeutici.
Sono attualmente in atto ulteriori ricerche basate su zebrafish per generare nuovi modelli malattia nei quali testare l’effetto di nuovi farmaci e/o di valutare l’impatto di molecole conosciute su nuovi possibili target terapeutici.
Sul fronte oncologico, per il quale la correlazione tra l’alterazione dell’espressione genica dipendente dagli RNA non-coding e lo sviluppo/progressione del cancro è ormai ben documentata, è in atto lo studio del ruolo di microRNA soppressori tumorali (TS-miRNA) nel carcinoma prostatico (PCa), alterandone l’espressione in linee cellulari di PCa in vitro. Al fine di ricostruire la rete di regolazione genica modulata da tali miRNA, vengono utilizzate tecniche per la cattura dei relativi target molecolari su larga scala, agevolandone l’identificazione (targetOME) tramite opportuno sequenziamento di nuova generazione (NGS). Si intende inoltre utilizzare i miRNA circolanti come biomarcatori predittivi nel PCa. In particolare sono state generate diverse linee cellulari di PCa in vitro resistenti al docetaxel, un farmaco usato quando il tumore raggiunge lo stadio di androgeno-indipendenza, per identificare i miRNA specificamente rilasciati da queste linee e quindi putativi biomarcatori di resistenza al farmaco.
Genetica e genomica di lievito
Il Saccharomyces cerevisiae, un organismo eucariotico unicellulare, è utilizzato come sistema genetico modello, poco costoso, di facile manipolazione, e a crescita assai veloce, per identificare interattori di proteine coinvolte nelle malattie e sviluppare nuovi saggi funzionali per la caratterizzazione delle mutazioni di geni umani e per dare un contributo per nuove terapie.
Le linee di ricerca sviluppate dal gruppo sono:
- Espressione eterologa di proteine coinvolte in malattie umane, screening a livello genomico e screening di librerie di cDNA per identificare interattori funzionali di proteine coinvolte nel cancro al seno e nel melanoma
- questa linea di ricerca si basa sul fatto che molti geni associati a malattie (il 50%) hanno un omologo in lievito e quindi, S. cerevisiae può essere un potente strumento per l'identificazione di interattori funzionali di queste proteine. L’ obiettivo è identificare e caratterizzare gli interattori funzionali di PARP-1 e BRAFV600E.
- questa linea di ricerca si basa sul fatto che molti geni associati a malattie (il 50%) hanno un omologo in lievito e quindi, S. cerevisiae può essere un potente strumento per l'identificazione di interattori funzionali di queste proteine. L’ obiettivo è identificare e caratterizzare gli interattori funzionali di PARP-1 e BRAFV600E.
- Sviluppo di test funzionali per caratterizzare mutazioni associate al cancro al seno
- un "test di ricombinazione in lievito" per valutare l’impatto funzionale di nuove varianti mis-senso BRCA1/2 che può fornire informazioni sulla biologia del tumore e sulla risposta terapeutica.
- un "test di ricombinazione in lievito" per valutare l’impatto funzionale di nuove varianti mis-senso BRCA1/2 che può fornire informazioni sulla biologia del tumore e sulla risposta terapeutica.
- Assemblaggio di vettori per la terapia genica in S. cerevisiae
- la terapia genica, la sostituzione o l'inserimento di una copia corretta di un gene difettoso, è una grande promessa per il trattamento delle malattie ereditarie, del cancro e delle infezioni croniche. Una limitazione importante all’uso di vettori virali in terapia genica è il costo della produzione. Al fine di ridurre i costi di produzione, si sta esplorando la fattibilità di produrre nel lievito un vettore virale ricombinante basato sul virus adeno-associato (rAAV).
- la terapia genica, la sostituzione o l'inserimento di una copia corretta di un gene difettoso, è una grande promessa per il trattamento delle malattie ereditarie, del cancro e delle infezioni croniche. Una limitazione importante all’uso di vettori virali in terapia genica è il costo della produzione. Al fine di ridurre i costi di produzione, si sta esplorando la fattibilità di produrre nel lievito un vettore virale ricombinante basato sul virus adeno-associato (rAAV).
Oncogenomica del melanoma
Le attività di Ricerca svolte, che nascono dalla sinergia tra il Core Research Laboratory dell’Istituto per lo Studio, la Prevenzione e la Rete Oncologica (ISPRO) e il CNR-IFC, si concentrano sullo studio del melanoma, la forma più aggressiva di cancro della pelle. Si intendono approfondire le conoscenze sulla regolazione e il funzionamento di BRAF, un oncogene che è molto frequentemente mutato in melanoma e rappresenta uno degli agenti eziologici di questa patologia.
Si stanno attualmente studiando i regolatori codificanti e non-codificanti dell’espressione di BRAF. Particolare attenzione è rivolta ai microRNA e ai competing endogenous RNA (ceRNA) che agiscono a livello post-trascrizionale. Sono anche sotto studio gli interattori della proteina BRAF, nonché gli effettori codificanti e non-codificanti della sua attività chinasica. I sistemi modello usati sono molteplici e vanno dall’in vitro (linee cellulari di melanoma, lievito S. cerevisiae) all’in vivo (topi immunocompromessi; zebrafish e topi geneticamente modificati). Lo scopo ultimo è identificare nuovi farmaci/target farmacologici da combinare con gli inibitori di BRAF che sono attualmente disponibili, per aumentarne l’efficacia.
Sono anche attive linee di ricerca per l’identificazione di microRNA che possano essere usati come biomarcatori predittivi di risposta e resistenza nei pazienti con melanoma metastatico sottoposti alla terapia mirata o all’immunoterapia.
Infine, si sta sperimentando in vivo una strategia innovativa che consente di veicolare selettivamente farmaci di varia natura all’interno delle cellule di melanoma.
Oligonucleotidi e Bioconiugazioni
Gli Oligonucleotidi (brevi sequenze sintetiche di DNA o RNA naturale o modificato) sono uno degli strumenti più importanti della biologia molecolare e delle biotecnologie. Infatti, possono essere utilizzati come sonde per riconoscere e legare acidi nucleici complementari o altre "biomolecole" alle quali si adattano; inoltre possono essere strumenti per “spegnere” o “regolare” l’espressione genica. A tale scopo, le principali attività di ricerca attuate sono rivolte alla progettazione, sintesi e purificazione di Oligonucleotidi per l'inibizione specifica delle funzioni cellulari, grazie all’interazione con RNA (siRNA, ribozomi e antisenso) con proteine (decoys, aptamers). In laboratorio gli studi sono rivolti allo sviluppo di aptameri mediante evoluzione in vitro da una popolazione oligonucleotidica randomizzata; e all'immobilizzazione/coniugazione e marcatura di biomolecole per l'imaging molecolare e il targeting di specifici bersagli molecolari.
La coniugazione di biomolecole (proteine, peptidi, acidi nucleici) con farmaci, fluorofori o altre "etichette", nanoparticelle o superfici solide di sensori, è un’altra biotecnologia, che consente la rilevazione, l’imaging e anche effetti terapeutici. È presente anche un expertise di derivatizzazione di superfici metalliche o siliconiche con strati organici e la preparazione di film funzionali di proteine/oligo per i biosensori. Il supporto "tecnologico" del laboratorio è rappresentato da strumenti per la sintesi oligonucleotida (DNA/RNA synthesizer), la purificazione di bioconiugati (HPLC e altre tecniche cromatografiche), e l'analisi (Capillary Electrophoresis), che, uniti all'esperienza chimica del personale, sono disponibili anche per ulteriori applicazioni e collaborazioni.
ProteOMICa
Il focus è la ricerca traslazionale, e in particolare l’applicazione di tecnologie di proteomica/spettrometria di massa e studi di biologia cellulare e a problematiche cliniche. Si mira ad ottenere nuove conoscenze sui meccanismi coinvolti nell’insorgenza e progressione di patologie, per identificare possibili biomarcatori prognostici e diagnostici (biomarker discovery) nonché definire e validare target molecolari per interventi terapeutici (proteomica funzionale). Da anni POL è impegnato nella ricerca delle basi molecolari di malattie cardiovascolari, metaboliche, oncologiche e genetiche/congenite sfruttando modelli cellulari e animali.
In particolare:
- l’analisi delle Cellule Muscolari Lisce Vascolari (VSMCs) ha reso possibile suggerire numerose proteine coinvolte nell’attivazione di queste cellule e quindi del ruolo che esse hanno nella crescita ed eventuale rottura delle placche aterosclerotiche;
- l’analisi della Matrice Extracellulare (ECM), oltre che di molecole infiammatorie, proteine secrete o rilasciate tramite microvescicole, intende individuare i ruoli delle varie componenti proteiche nell’aterogenesi e nella progressione e complicazione della patologia;
- l’analisi del proteoma e secretoma dei fibroblasti in un modello cellulare di fibrosi polmonare viene svolta nell’ambito dello studio del proteoma mitocondriale in un modello murino di ischemia cardiaca e di obesità.
- lo studio del proteoma salivare intende ottenere nuove conoscenze in patologie croniche (come la Leucemia Mieloide Cronica – CLM) e sistemiche/infiammatorie (Sindrome di Sjogren).
- lo studio di disordini genetici di accumulo lisosomiale (CLN1, CLN5) è portato avanti in collaborazione con Stella Maris e la Scuola Normale Superiore, utilizzando tecnologie di proteomica/lipodimica e sfruttando nanostrutture per la veicolazione intracellulare di farmaci ed enzimi.
POL ospita una piattaforma proteomica completa provvista di spettrometri di massa e attrezzature per gel elettroforesi mono e bi-dimensionale e cromatografie; tre spettrometri di massa costituiscono il nucleo centrale e più prezioso del laboratorio: 5600 Triple TOF, 5500 QTrap and 4800 MALDI TOF TOF.
|
Staff laboratorio FG2 per unità di Ricerca |
||||
|
Non-coding RNA |
Genetica e Genomica di lievito |
Oncogenomica del melanoma |
Oligonucleotidi e BioConjugation |
ProteOMICs Lab. (POL) |
|
Letizia Pitto Milena Rizzo |
Alvaro Galli Tiziana Cervelli |
Laura Poliseno |
Lorena Tedeschi |
Silvia Rocchiccioli Antonella Cecchettini |




 中文
中文  EN
EN